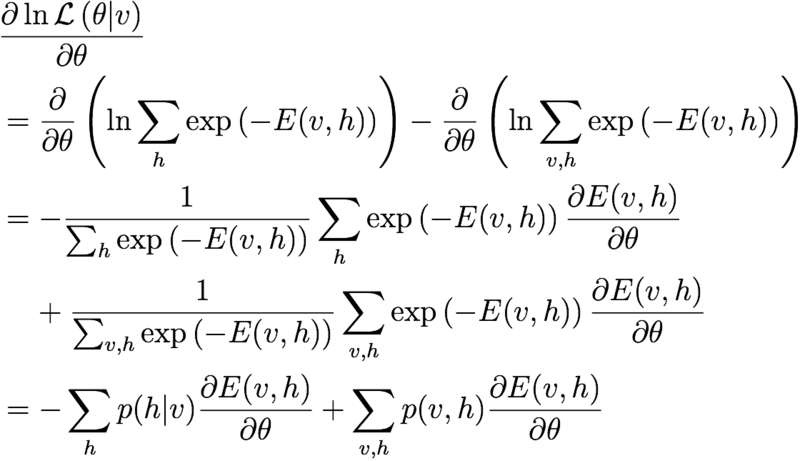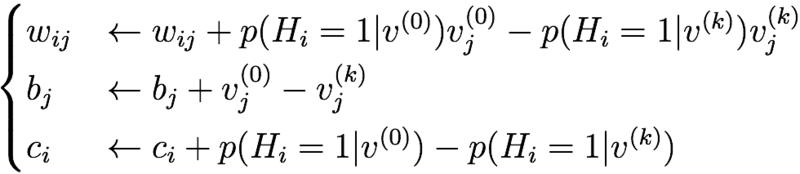ゼロから始めるDeepLearning_その4_Restrictedボルツマンマシンとは
はじめに
MNISTで全然うまく行かないことが発覚したので、最適化を調べ中きちんと動きました。
学部四年生向け。だった。
ニューラルネットワーク
→(AutoEncoder)
→(DenoisingAutoEncoder)
→ホップフィールドネットワーク
→ボルツマンマシン
→Restrictedボルツマンマシン(この記事)
→(Gaussian Binary - Restricted Boltzmann Machines)
→(DeepBeliefNetwork)
→(DeepNeuralNetworks)
→畳み込みニューラルネット(後日)
太線以外は読み飛ばしてOK
Restricted Boltzmann Machines(RBM)をとりあえず使ってみる
RBMには、ホップフィールドネットワークという前身がある。
できることは、それと同じである。
即ち、RBMとは、脳的なもの(マルコフ確率場)に、画像を複数覚えさせるものである。
1.画像を用意する(ノイズなしの元画像)
覚えさせる画像その2(CameraMan:111*111画素)

2.画像から結合係数wを学習する
焼きなまし法を用いて、上の2つの画像より、以下の結合係数wを学習する。
(ここでは4例を上げるが、実際には30*(111*111)の行列で、30例を上げることができる)
(結合係数wには、入力画像に近しいものがいくつもあるが、この理由は後述する。)
3.学習した結合係数wを用いて、ノイズ入り画像から元画像を復元する
ノイズ入り画像とは、以下の様なものである。
(ぱっと見、結合係数wと似てるけど別物。元画像+ノイズによって生成)
これをRBMの入力層→隠れ層→出力層(実際には入力層と同じ)と変換すると、ノイズなし画像が復元される。
実際に、結合係数wを用いると、ノイズ入りの画像から、以下の様な画像を復元することができる。
これらは、学習途中の結合係数wによるものである。
もっと学習回数を重ねた結合係数wを用いると、以下のような画像が復元される。
最終的には、鮮明な画像を復元可能な結合係数wが学習され、以下のように復元される。
概要・Restricted ボルツマンマシンってなに?
DeepLearning(DeepBeliefNetworks)で使われるのは、
Restricted Boltzmann machine(接続制限されたボルツマンマシン)と呼ばれ、
前章のホップフィールドネットワークの改良であるボルツマンマシンの特殊な事例と言える。
図的に書くと、以下の様なものである。
1.図中でvと書かれている可視素子(入出力を行う素子:visible unit)に、
{0,1}の2値で表現可能な入力画像を入れる。
2.可視素子vが結合係数wによって、隠れ素子h(hidden unit)に入力され、
その入力にしたがって生成される確率分布に従って、隠れ素子hが{0,1}の値を取る。
(σはシグモイド関数、cはバイアス)
3.今度は逆に、隠れ素子hが結合係数wによって、可視素子vに入力され、
その入力にしたがって生成される確率分布に従って、可視素子vが{0,1}の値を取る。
(この可視素子の出力が入力した元画像に一致することを期待している。)
(bはバイアス)
4.以上の工程から得られる確率が入力したvの分布に近づくように
パラメータを学習する。
(はパラメータw,b,cのこと)
5.学習し終わったRBMに元画像にノイズを加えた画像を入力すると、
ノイズがなくなった元画像が出力される。
あえてニューラルネット的に書くと、こんな感じになる。
この手法が作られたのは、以下の経緯による。
で表される入力画像は、
通り考えうる。
(入力は空間上のある分布から生成されている)
素子数をnとすると、ボルツマンマシンのパラメータの自由度はn(n+1)/2である。
(結合係数wijとしきい値θi)
ボルツマンマシンは空間中で、なおかつモデルの制約を満たす1つの分布を表現しているにすぎない。
(モデルが導入されているので、導入した構造によって自由度が下がっている)
この自由度の差を埋めるために、m個の隠れ素子という概念を導入する。
入出力用の素子はある意味、決まった値を取らなければならない。
隠れ素子にはそのような制約がなく、モデルを複雑化することができる。
隠れ素子の導入によってボルツマンマシンの自由度は(n + m)(n + m + 1)/2にまで増加する。
しかし、こういった素子を増やすと、過学習などが発生して大変なことになる。
ついでに学習コストも増大する。
しかたがないので、素子の接続をある程度制限することで、これらの問題を回避しようとした。
その結果生まれたのが、接続制限されたボルツマンマシン(RBM)である。
またこのRBMにおいて有名な最適化手法が、Hintonの作った
Contrastive divergence法というものであり、
これによって現実的なデータサイズに対する十分実用的な学習ができるようになった。
理論・Restricted ボルツマンマシン
以下、内容を説明するために色々な式が飛び交います。
実装だけできれば良い場合は、本ページ末尾にあるRのコードを写すか、
An Introduction to Restricted Boltzmann Machinesの28ページを読んで下さい。
ホップフィールドネットワークと同様に、エネルギー関数(評価関数)を定義する。
(隠れ素子がhとして分離されているだけで、同じ式)
このエネルギー関数は、可視素子
と隠れ素子
の積(相関)と、
結合係数が一致する時に、最も小さな値(最尤値)を取る。
(結合係数wには、入力画像に近しいものがいくつもあるのは、このエネルギー関数による)
またボルツマンマシンと同様に、素子の発火確率pの式を定義する。
具体的には、以下の様に、
したがって、各パラメータごとの勾配は以下のようになる。
よって、以下の更新を繰り返せば良い。
しかしここで問題となるのが、という項である。
とは、入力画像としてありうる{0,1}のパターン全体のことである。
つまり、指数オーダーで組み合わせ爆発を起こす確率分布を、
実際には存在しないデータも含めて計算しろと言われている。無理。
ここで普通は、十分長いギブスサンプリングを繰り返してを近似することを考える。
ただ、それには問題がある。
(以下Hinton, Geoffrey E.の翻訳)
ギブスサンプリングでは、確かに平衡分布を計算可能ではあるが、
そのサンプリングは、モデルが形成しうる分布の至るところから行われるため、一般に大きな分散を持つ。
この分散は、変な局地を作る。
また、このサンプリング結果は、モデルのパラメータにも依存する。
したがって、例えあるパラメータを取る時、勾配が0になるとしても、
サンプリングにおいて大きな分散を持つならば、そのパラメータは弾かれてしまう。
(所々が振動している平らな板に砂をばらまくと、振動していないところに砂が集まるイメージ。
今回問題となるのは、振動しているところが、実は時間平均を取ると、振動していない(勾配0)場合)
なので、Hintonは別のやり方を考えた。
Contrastive Divergence法である。
(Hinton, Geoffrey E. "Training products of experts by minimizing contrastive divergence." )
ある入力ベクトル(一枚の画像)をとする。
入力ベクトルを用いてk回後のサンプリングで得られたある1つの入力(出力)ベクトルをとする。
(このkは基本的に1で良い)
この2つを用いて、十分長いギブスサンプリングを繰り返した後のを近似すると、
対数尤度関数の微分は以下のようになる。
この手法は、平衡状態に至るまでギブスサンプリング(MCMC法)を用いて、
最初状態と最後の状態の導関数(微分)を比較するのではなく、
1ステップのマルコフ連鎖(サンプリング)によって、パラメータを更新する。
これによって、マルコフ連鎖にありがちな、最初の更新で初期分布から離れてさまよってしまう傾向を減らしている。
ざっくり言うと、この勾配が0になるのは、モデル(結合係数w)が完全な時のみなので、
この更新を繰り返して勾配がなくなったところを見れば十分という理屈。
したがって画像一枚ごとに、以下の更新を繰り返せば良い。
終了。
ちなみに、一番良くわからない隠れ層の素子数だが、
の空間における任意の分布は、
覚えさせる画像の数+1個の隠れ素子を用いることで近似できるらしい。
(Le Roux, Nicolas, and Yoshua Bengio.
"Representational power of restricted Boltzmann machines and deep belief networks." )
直感的には、結合係数wが(画像の種類×画像のピクセル数)の大きさを持っていれば、
結合係数w自体を画像そのものにすることで、すべての画像を記録できるためと思われる。
(冒頭の実験で、結合係数wが画像データになっているのは、そういう理屈)
この理論はDeepでも頻繁に出てくるので、わりと重要。
実装・R
2014/03/08 引数を修正2014/03/12 細かい修正
#使い方 if(FALSE){ library('bmp') data1 <- read.bmp('Downloads/mono/LENNA.bmp') data2 <- read.bmp('Downloads/mono/Cameraman.bmp') #実際には大きすぎてメモリが足りないので #dataの一部を小さく切り出してください data <- rbind(c(data1), c(data2)) tmp <- list(NULL) tmp$w <- NULL #学習 for(i in 1:10){ tmp <- rbm(train.mat=data,h.num=30,iter.num=10,w.mat=tmp$w,b.vec = tmp$b, c.vec = tmp$c) } #復元 huge <- list(NULL) for(i in 1:2){ huge[[i]] <- tmp$w %*% as.vector(data[i,] + rnorm(mean = 0, sd = 0.5, n = 111^2)) + tmp$c huge[[i]] <- 1 / ( 1 + exp( -huge[[i]] )) huge[[i]] <- t(tmp$w) %*% huge[[i]] + tmp$b huge[[i]] <- 1 / ( 1 + exp( -huge[[i]] )) image(matrix(huge[[i]], nrow=111)) } } #Restricted Boltzmann Machines rbm <- function(train.mat, h.num = 10, b.vec = NULL, c.vec = NULL, w.mat = NULL, iter.num = 2, learn.rate = 0.001){ #Sigmoid sigmoid <- function(x){ return( 1/(1+exp(-x)) ) } #Contrastive Divergence cd <- function(b.vec, h.vec, c.vec, w.mat, train.mat, iter.num = 2){ #勾配の初期化 dw.mat <- matrix(0, nrow = dim(w.mat)[1], ncol = dim(train.mat)[2]) dc.vec <- rep(0,dim(w.mat)[1]) db.vec <- rep(0,dim(train.mat)[2]) h.vec <- rep(0,dim(w.mat)[1]) dev.next() par(mar=c(0,0,0,0)) layout(matrix(1:(3*dim(w.mat)[1]*dim(train.mat)[1]),nrow = 3)) for(l in 1:dim(train.mat)[1]){ v.init.vec <- train.mat[l,] v.vec <- train.mat[l,] #step.vec <- rep(1,iter.num) step.vec <- seq(from=1000,to=1,length.out = iter.num+1) #初回のみhは0,1あとは確率値 #h.vec p1 <- (w.mat %*% v.vec + c.vec) #p1[-which.max(p1)] <- 0 #p1 <- (p1 - mean(p1))/sd(p1) p1 <- sigmoid( p1 ) h.vec <- rbinom(n=length(h.vec),size=1,prob=p1) #v.vec p1 <- ( t(w.mat) %*% h.vec + b.vec) #p1 <- (p1 - mean(p1))/sd(p1) p1 <- sigmoid( p1 ) v.vec <- p1 #calc grad pv0 <- sigmoid( ( w.mat %*% v.init.vec + c.vec) ) pvk <- sigmoid( ( w.mat %*% v.vec + c.vec) ) for(i in 1:length(pv0)){ image( matrix( (pv0 %*% c(v.init.vec))[i,], 28 ) ) image( matrix( w.mat[i,] , 28)) image( matrix( (pvk %*% c(v.vec))[i,], 28 ) ) } dw.mat <- dw.mat + pv0 %*% c(v.init.vec) - pvk %*% c(v.vec) dc.vec <- dc.vec + pv0 - pvk db.vec <- db.vec + v.init.vec - v.vec } ans <- list(dw.mat, db.vec, dc.vec) names(ans) <- c('dw','db','dc') return(ans) } dims.vec <- c(dim(train.mat)[2], h.num) if(is.null(w.mat)){ w.mat <- matrix(rnorm(mean=0,sd=0.001,n=prod(dims.vec)), nrow = dims.vec[2], ncol = dims.vec[1]) } p1 <- sigmoid( (w.mat%*% train.mat[1,] + 0) ) if(is.null(c.vec)){ c.vec <- log(abs((p1+0.01)/(1-p1))) # c.vec <- runif(min=-0.01,max=0.01,n=dims.vec[2]) } p1 <- sigmoid( (t(w.mat)%*%p1 ) ) if(is.null(b.vec)){ b.vec <- log(abs((p1+0.01)/(1-p1))) # b.vec <- rnorm(mean=0,sd=0.01,n=dims.vec[1]) } h.vec <- rep(0,dims.vec[2]) for(i in 1:dim(train.mat)[1]){ idx <- c(1:h.num) #idx <- sample(c(1:h.num),size=round(0.5 * h.num),replace=FALSE) hoge <- cd(b.vec,h.vec[idx],c.vec[idx],w.mat=w.mat[idx,],matrix(train.mat[i,],ncol=dim(train.mat)[2]),iter.num) w.mat[idx,] <- w.mat[idx,] + learn.rate * hoge$dw b.vec <- b.vec + learn.rate * hoge$db c.vec[idx] <- c.vec[idx] + learn.rate * hoge$dc } ans <- list(w.mat, b.vec, c.vec) names(ans) <- c('w','b','c') return(ans) } recover.rbm <- function(data.mat = NULL,para = NULL){ dev.next() par(mar=c(0,0,0,0)) layout(matrix(1:40,nrow = 4)) idx.vec <- list(sample(c(1:dim(data.mat)[1]),size=min(c(dim(data.mat)[1],10)),replace=FALSE), sample(c(1:dim(para$w)[1]),size=min(c(dim(para$w)[1],10)),replace=FALSE)) huge <- list(NULL) #for(i in 1:min(c(dim(data.mat)[1],10))){ for(i in 1:10){ image(matrix(data.mat[idx.vec[[1]][i],], nrow=sqrt(dim(data.mat)[2]))) image(matrix(para$w[idx.vec[[2]][i],], nrow=sqrt(dim(data.mat)[2]))) huge[[i]] <- para$w %*% data.mat[idx.vec[[1]][i],] + para$c huge[[i]] <- 1 / ( 1 + exp( -(huge[[i]]) )) huge[[i]] <- (t(para$w) %*% huge[[i]]) + para$b image(matrix(huge[[i]], nrow=sqrt(dim(data.mat)[2]))) huge[[i]] <- 1 / ( 1 + exp( -(huge[[i]]) )) image(matrix(huge[[i]], nrow=sqrt(dim(data.mat)[2]))) } }